Separazione di proteine e peptidi. Metodi per la separazione di proteine e amminoacidi Metodi per la separazione di proteine
Lo studio delle proprietà fisico-chimiche, della composizione chimica e della struttura è possibile solo studiando una preparazione proteica purificata. Per isolare e frazionare le singole proteine si utilizzano: salatura, precipitazione con solventi organici, filtrazione su gel, elettroforesi, cromatografia a scambio ionico, cromatografia di affinità.
Salare le proteine basato sulla dipendenza della solubilità delle proteine dalle proprietà del mezzo. Le proteine sono meno solubili in acqua distillata che in soluzioni saline deboli, poiché basse concentrazioni di ioni mantengono i loro gusci di idratazione. Ma ad alte concentrazioni di sale, le molecole proteiche perdono i loro gusci di idratazione, si aggregano e si forma un precipitato. Dopo che il sale è stato rimosso, le proteine ritornano in soluzione, mantenendo le loro proprietà e conformazione originarie.
I cambiamenti nella solubilità a diverse concentrazioni di sale e pH vengono utilizzati per isolare le singole proteine. Molto spesso, per salare le proteine vengono utilizzate soluzioni di solfato di ammonio di diverse concentrazioni.
La precipitazione delle proteine dalla soluzione senza la loro denaturazione viene effettuata utilizzando agenti deidrogenanti - solventi organici (etanolo, acetone).
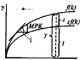
Filtrazione su gel basato sulla separazione delle proteine in base alla dimensione e alla forma della molecola. La separazione viene effettuata in colonne cromatografiche riempite con granuli di gel poroso (Sephadex, agarosio) in una soluzione tampone con un determinato valore di pH. I granuli di gel sono permeabili alle proteine grazie a canali interni (pori) di un certo diametro medio, la cui dimensione dipende dal tipo di gel (Sephadex G-25, G-200, ecc.). La miscela proteica viene aggiunta alla colonna e poi lavata (eluita) con una soluzione tampone con un determinato valore di pH. Le grandi molecole proteiche non penetrano nei pori del gel e si muovono ad alta velocità insieme al solvente. Piccole molecole di impurità a basso peso molecolare (sale) o altre proteine vengono trattenute dai granuli di gel e vengono lavate via dalla colonna più lentamente (Fig. 1.29). All'uscita della colonna la soluzione (eluato) viene raccolta sotto forma di frazioni separate.
Riso. 1.29. Separazione delle proteine mediante filtrazione su gel
Elettroforesi si basa sulla proprietà delle molecole proteiche cariche di muoversi in un campo elettrico ad una velocità proporzionale alla loro carica totale. Le proteine che hanno una carica totale negativa ad un dato valore di pH si muovono verso l'anodo, mentre una carica positiva si muove verso il catodo. L'elettroforesi viene eseguita su diversi supporti: carta, gel di amido, gel di poliacrilammide, ecc. La velocità di movimento dipende dalla carica, dalla massa e dalla forma delle molecole proteiche. Dopo il completamento dell'elettroforesi, le zone proteiche sul supporto vengono colorate con coloranti speciali (Fig. 1.30, A).
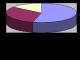
La risoluzione dell'elettroforesi in un gel è maggiore che su carta, quindi quando si esegue l'elettroforesi delle proteine del siero del sangue su carta, vengono isolate 5 frazioni (albumina, α 1 -, α 2 -, β-, γ-globuline) e in un gel di poliacrilammide - fino a 18 frazioni (Fig. 1.30, B).
Riso. 1.30. Elettroferogramma delle proteine del siero del sangue di una persona sana
UN- elettroferogramma delle proteine del siero del sangue su carta;
B- la quantità di proteine plasmatiche di diverse frazioni.
I - γ-globuline; II - β-globuline; III - a 2 -globuline;
IV - a 1 -globuline; V - albumine
Cromatografia a scambio ionico basato sulla separazione di proteine che differiscono nella carica totale. Una soluzione proteica con un determinato valore di pH viene fatta passare attraverso una colonna cromatografica riempita con un assorbente solido poroso, mentre alcune proteine vengono trattenute a seguito dell'interazione elettrostatica. Le sostanze a scambio ionico vengono utilizzate come assorbenti: scambiatori di anioni (contenenti gruppi cationici) per isolare le proteine acide; scambiatori cationici (contenenti gruppi anionici) per isolare le proteine essenziali.
Quando si fa passare una proteina attraverso una colonna, la forza del suo legame con lo scambiatore ionico dipende dall'entità della carica opposta alla carica dell'assorbente. Le proteine adsorbite su un adsorbente a scambio ionico vengono eluite con soluzioni tampone con diverse concentrazioni saline e pH, ottenendo diverse frazioni proteiche.
Cromatografia di affinità si basa sulla specificità di legame della proteina al ligando attaccato al supporto solido. Come ligandi vengono utilizzati substrati enzimatici, gruppi protesici di oloproteine, antigeni, ecc. Quando si fa passare una miscela di proteine attraverso la colonna, solo la proteina complementare si attacca al ligando (Fig. 1.31, A), tutte le altre escono insieme alla soluzione. La proteina adsorbita viene eluita con una soluzione con un valore di pH diverso (Fig. 1.31, B). Questo metodo è altamente specifico e consente di ottenere preparati proteici altamente purificati.
L'isolamento e la purificazione delle proteine avvengono solitamente in più fasi utilizzando metodi diversi. La sequenza dei passaggi è selezionata empiricamente e può variare per proteine diverse. Un elevato grado di purificazione delle proteine è molto importante sia quando vengono utilizzate come medicinali (l'ormone insulina, ecc.), sia quando si diagnosticano varie malattie modificando la composizione proteica dei tessuti, del sangue, della saliva, ecc.
L'insieme delle proteine nelle cellule dei vari organi di un adulto è individuale e viene mantenuto relativamente costante per tutta la vita. I tessuti specializzati possono contenere proteine specifiche, come l’emoglobina nei globuli rossi, l’actina e la miosina nei muscoli, la rodopsina nella retina e diversi tipi di collagene nelle ossa e nei tessuti connettivi. Alcune proteine si trovano in molti tessuti, ma in quantità diverse. La composizione selezionata cambia
Riso. 1.31. Separazione delle proteine mediante cromatografia di affinità
UN- legame della proteina isolata ad un ligando specifico attaccato ad un trasportatore neutro; B- ottenere una soluzione di proteine individuali
le proteine dei tessuti e del sangue sono possibili e sono associate principalmente alla dieta, alla composizione del cibo e all'attività fisica di una persona.
Nelle malattie, la composizione proteica delle cellule del sangue e dei tessuti può cambiare in modo significativo, spesso si sviluppa una carenza di qualsiasi proteina o una diminuzione della sua attività; proteinapatia. Pertanto, la determinazione di cambiamenti pronunciati nella composizione proteica del sangue e dei tessuti viene utilizzata per diagnosticare varie malattie negli studi clinici.
Lo studio dell'omogeneità dei preparati proteici e l'isolamento delle singole frazioni proteiche viene effettuato utilizzando vari metodi, i più importanti dei quali si basano sull'uso dell'ultracentrifugazione, dell'elettroforesi, della cromatografia, nonché sullo studio della solubilità proteica.
1. Metodi per separare proteine e amminoacidi in base alle differenze di peso molecolare delle sostanze:
a) ultracentrifugazione. In un'ultracentrifuga vengono precipitate per prime le molecole più pesanti, seguite da quelle meno pesanti.
b) filtrazione su gel. Con questo metodo, una colonna cromatografica viene riempita con granuli porosi di un polimero di carboidrati altamente idratato, molto spesso Sephadex (derivati appositamente trattati del destrano di carboidrati ad alto peso molecolare). Quando si filtra una miscela di proteine a basso e ad alto peso molecolare attraverso tale colonna, le piccole molecole proteiche, penetrando attraverso i pori all'interno dei granuli Sephadex, fluiranno attraverso la colonna più lentamente delle proteine le cui molecole non si adattano i pori dei granuli e quindi fuoriescono più velocemente dalla colonna.
2. Metodi per separare proteine e amminoacidi in base alle differenze nelle loro proprietà acido-base (o alle differenze nelle loro cariche elettriche):
a) metodo dell'elettroforesi. Lo scopo dell'elettroforesi è separare le sostanze in soluzione in un campo elettrico in base alle differenze nelle loro cariche elettriche. Gli studi elettroforetici sulle proteine vengono solitamente eseguiti a diversi valori di pH, perché È stato stabilito che se ad un pH una preparazione proteica si comporta come una sostanza omogenea, ad un altro pH la stessa preparazione può essere eterogenea.
Negli ultimi anni si è diffusa l'elettroforesi di soluzioni di proteine e peptidi su vari mezzi: carta da filtro, polvere di cellulosa o amido, gel di poliacrilammide. Questi metodi consentono l'analisi di quantità estremamente piccole di proteine.
b) elettroforesi su disco in gel di poliacrilammide, in cui una miscela di proteine viene esposta contemporaneamente ad un campo elettrico e ad un gradiente di pH. Ha una risoluzione particolarmente elevata.
La filtrazione su gel, così come l'elettroforesi su gel di poliacrilammide, è ampiamente utilizzata per stimare rapidamente il peso molecolare delle proteine.
c) cromatografia a scambio ionico. Nella cromatografia a scambio ionico, i polimeri che trasportano una carica vengono utilizzati come trasportatori - resine a scambio ionico:
· resine a scambio cationico (caricate negativamente) – scambiano cationi;
· resine a scambio anionico (caricate positivamente) – scambiano anioni.
Ad esempio, viene spesso utilizzata una resina polisteroide solfonata a scambio cationico. Se la soluzione di amminoacidi è acida, quando la colonna viene caricata, gli amminoacidi e le proteine caricati positivamente sostituiscono il sodio e si combinano con l'anione solfuro. Quando viene aggiunto idrossido di sodio, il pH aumenta; Quando il pH raggiunge un valore pari al punto isoelettrico della molecola proteica, gli aminoacidi perdono la loro carica e diventano neutri. Sotto l'influenza della gravità, l'amminoacido lascia la colonna avendo perso la sua carica. Diverse proteine (amminoacidi) hanno diversi valori di punti isoelettrici.
3. Metodi di separazione basati sulle differenze di solubilità delle sostanze:
a) metodo di frazionamento delle proteine con soluzioni saline. Si basa sul fatto che ogni singola proteina della miscela da separare viene precipitata da essa ad una certa concentrazione di un particolare sale, mentre altre proteine ad una data concentrazione di sale rimangono in soluzione. Il processo di precipitazione delle proteine dalla soluzione sotto l'influenza del sale è chiamato salatura. Con un'ulteriore saturazione con sale, la proteina individuale successiva scompare e così è possibile isolare una dopo l'altra proteine individuali relativamente pure.
b) cromatografia di partizione su carta. Questo metodo si basa su diversi gradi di distribuzione dei componenti della miscela tra due fasi liquide immiscibili (mobile e stazionaria) e consiste nel porre una goccia di idrolizzato proteico su una striscia di carta cromatografica, un'estremità della quale viene immersa in una soluzione organica solvente. Il solvente viene assorbito dalla carta sotto l'azione delle forze capillari e, passando lungo una striscia di carta, trasporta con sé gli amminoacidi.
La velocità con cui gli amminoacidi si muovono attraverso la carta dipende dalla loro struttura chimica e dalla capacità di dissolversi in solventi mobili e stazionari. Come solvente mobile vengono utilizzati fenolo saturo di acqua, alcol n-butilico, ecc. Il solvente stazionario è l'acqua, i cui vapori saturano la carta. Minore è la solubilità degli amminoacidi in acqua e maggiore la loro solubilità, ad esempio nel fenolo, più velocemente si spostano dietro il fronte del solvente organico.
4. Determinazione della struttura primaria della proteina
La procedura più importante per stabilire la struttura primaria delle proteine è determinare la sequenza dei residui aminoacidici. Attualmente, questo lavoro viene svolto principalmente utilizzando il metodo del fenilisotiocianato di Edman.
Il metodo Edman è implementato in un dispositivo creato appositamente per questo scopo, chiamato sequenziatore (da sequenza). Il metodo Edman prevede il trattamento di una proteina o peptide attaccato tramite un amminoacido C-terminale a un supporto inerte (polistirene o vetro poroso) in una colonna sequenziatore con fenil isotiocianato. Dopo aver lavato la colonna con solventi (metanolo, dicloroetano), il feniltiocarbamil peptide risultante viene esposto all'acido trifluoroacetico anidro, a seguito del quale vengono rilasciati anilinotiozolinone e il suo amminoacido N-terminale e il peptide o proteina, accorciato di un amminoacido residuo, resta vincolato al trasportatore.
Sezione 3. NUCLEOTIDI E ACIDI NUCLEICI
Lezione 4. Struttura e funzioni dei nucleotidi
1. Caratteristiche generali dei nucleotidi
Nucleotidi - sostanze organiche complesse costituite da 3 componenti essenziali:
1) base azotata;
2) zucchero a cinque atomi di carbonio;
3) residuo di acido fosforico.
I composti organici complessi costituiti solo da una base azotata e da uno zucchero pentoso sono chiamati nucleosidi. Pertanto, i nucleotidi sono esteri fosforici dei nucleosidi.
Basi azotate
Le basi azotate sono derivati di due composti eterociclici: purina e pirimidina:
Basi azotate purine:

Adenina Guanina
basi azotate pirimidiniche:

Uracile citosina timina
Cinque zuccheri di carbonio:


β-ribosio β-desossiribosio
Acido fosforico

La composizione dei nucleotidi include necessariamente un residuo di acido fosforico (ortofosforico).
Oltre ai tre componenti richiesti sopra, le molecole nucleotidiche possono contenere anche altri gruppi funzionali.
Durante la formazione dei nucleosidi, il primo atomo di ribosio (desossiribosio) si lega all'atomo N-1 della pirimidina o all'atomo N-9 della base purinica.
L'adenina si combina con il ribosio per formare adenosina; guanina, formando guanosina; citosina, formando citidina; uracile per formare uridina.
Adenina, guanina, citosina e timina si combinano con il desossiribosio per formare rispettivamente deossiadenosina, deossiguanosina, deossicitidina e timidina.
L'aggiunta più comune in natura è alla 5° posizione dello zucchero e non è indicata.
Nel corpo, i nucleotidi sono monomeri di acidi nucleici o funzionano in modo indipendente. A seconda della quantità dei loro componenti principali nei nucleotidi, tutti i nucleotidi sono suddivisi in mononucleotidi, dinucleotidi e polinucleotidi (acidi polinucleici).
2. Struttura e funzioni dei mono- e dinucleotidi
I mono- e i dinucleotidi non fanno parte degli acidi nucleici; funzionano in modo indipendente. I nucleotidi indipendenti contengono sempre ribosio come zucchero.
I mononucleotidi includono ATP, ADP, AMP, coenzima A e altri nucleotidi.
ATP – acido adenosina trifosforico:

L'ATP è l'equivalente energetico della cellula; è un intermediario tra le reazioni che rilasciano energia (esergoniche) e le reazioni che assorbono energia (ergoniche). In altre parole, la cellula immagazzina energia sotto forma di ATP, che viene poi utilizzata per i processi vitali.
I legami chimici tra diversi atomi nei composti organici sono divisi in 2 tipi:
1) normale
2) macroergico
Connessioni normali– legami, alla formazione o al decadimento dei quali la variazione del livello di energia libera dei composti è di 12,5 J/mol.
Connessioni macroergiche– legami, alla formazione o al decadimento dei quali il livello di energia libera del composto è di 25-50 kJ/mol della sostanza.
Il concetto di “legame macroergico” tiene conto dell'effetto energetico di un legame trasformato attraverso una reazione chimica di una sostanza con proprietà normali.
I legami tra i residui di acido fosforico sono ad alta energia: l'energia viene rilasciata durante la loro idrolisi. Tali connessioni sono solitamente indicate da una linea ondulata.
L'energia della prima molecola di ATP può servire solo per la prima reazione. ADP e AMP non sono in grado di essere una fonte di energia.
Esistono 3 modi per produrre ATP nelle cellule viventi:
1) Fosforilazione del substrato.
2) Fosforilazione ossidativa.
3) Fosforilazione fotosintetica.
Coenzima A (CoA). Il CoA è un trasportatore del gruppo acilico; partecipa a molti processi. Contiene adenina, ribosio, pirofosfato, acido pantotenico (vitamina B 3) e tiolamina. Semplificando, il coenzima A è rappresentato dalla seguente formula: HS-KoA. Quando il coenzima A interagisce con l'acido acetico, si forma l'acetil coenzima A, nella cui molecola appare un macroergico (ad alta energia):

Acetil coenzima A
L'acetil coenzima A è un metabolita chiave, grazie al quale avviene non solo la scomposizione e la sintesi di varie sostanze, ma anche la relazione tra i processi del metabolismo di proteine, lipidi e carboidrati.
I dinucleotidi includono NAD, NADP, FAD, ecc.
SOPRA - nicotinammide adenina dinucleotide;
NADP – nicotinammide adenina dinucleotide fosfato.
Questi dinucleotidi includono la nicotinamide (ammide dell'acido nicotinico, che è un'importante vitamina - vitamina B 5). La molecola NADP è identica nella struttura al NAD, con l'unica differenza che nel NADP, nell'atomo C-3 del ribosio, il gruppo OH è sostituito da un residuo molecolare di acido fosforico.
Le molecole NAD e NPDP sono capaci di ossidazione e riduzione reversibili (grazie alla capacità redox della nicotinamide), quindi partecipano come trasportatori di idrogeno; nelle reazioni di ossidazione biologica, NAD e NADP sono cofattori degli enzimi deidrogenasi.

Struttura del NAD (forma ossidata)
FAD – flavina adenina dinucleotide. Contiene riboflavina (vitamina B2).

Struttura del FAD (forma ossidata)
Il FAD, come altri dinucleotidi, è capace di ossidazione e riduzione reversibile aggiungendo 2 atomi di idrogeno alla sua molecola, quindi partecipa all'ossidazione biologica come trasportatore di idrogeno. È un cofattore delle deidrogenasi, nonché del NAD e del NADP.
3. Struttura e funzioni degli acidi nucleici
La proprietà più notevole delle cellule viventi è la loro capacità di riprodurre le proprie specie con una precisione quasi estrema e non solo una o due volte, ma in centinaia e migliaia di generazioni.
Le cellule viventi hanno questa capacità grazie alla presenza di acidi nucleici al loro interno.
DNA – acido desossiribonucleico;
L'RNA è l'acido ribonucleico.
Il DNA e l'RNA sono composti ad alto peso molecolare costruiti sulla base di nucleotidi collegati da legami 3, 5 fosfodiesteri. Il loro peso molecolare varia notevolmente (da 15mila a 1 miliardo).
Gli acidi nucleici sono altamente solubili nei fenoli; cattivo - in soluzioni di acidi deboli.
Differenze tra DNA e RNA:
1. Il DNA contiene adenina, guanina, citosina, timina;
L'RNA contiene adenina, guanina, citosina, uracile.
2. Il DNA contiene desossiribosio; L'RNA contiene ribosio.
3. Molecole di DNA a doppio filamento; L'RNA è a filamento singolo.
Caratteristiche della struttura del DNA
· Il DNA è costituito da due eliche polinucleotidiche destrorse che hanno un asse comune.
· I due filamenti del DNA sono antiparalleli, cioè I ponti fosfodiestere 3 e 5 sono orientati in direzioni opposte.
· Le basi sono piatte, idrofobe, situate su piani paralleli e perpendicolari all'asse lungo delle spirali.
· Le basi delle 2 catene sono accoppiate. Di fronte alla AT; opposto G-C;
Le basi accoppiate sono complementari tra loro.
Complementarità– complementarità spaziale delle superfici delle molecole interagenti o delle loro parti, portando all’emergere di legami secondari tra di loro.
Ci sono 2 legami idrogeno tra A e T; tra G e C – 3 legami idrogeno.
Residui di zucchero e gruppi fosforo rimangono sulla superficie della molecola e entrano in contatto con l'acqua. Gruppi caricati negativamente di residui di acido fosforico interagiscono facilmente con le proteine, tra le quali predominano gli istoni, proteine che differiscono nella loro natura basica.
4. Gli acidi nucleici differiscono tra loro nelle funzioni.
Le funzioni del DNA sono l'immagazzinamento, la replicazione (raddoppio) e la trasmissione delle informazioni ereditarie (le informazioni ereditarie sono informazioni sulla struttura primaria delle proteine).
Le funzioni dell'RNA sono determinate dal tipo di RNA.
Tipi di RNA:
a) m-RNA – matrice o i-RNA – informativo.
L'RNA messaggero svolge la funzione di trasferire le informazioni ereditarie dal nucleo cellulare dal DNA al citoplasma, al sito di sintesi proteica.
L'implementazione delle informazioni ereditarie è la sintesi proteica.
Ci sono centinaia di migliaia di tipi di m-RNA in una cellula.
b) t-RNA – trasporto.
Trasporta gli aminoacidi essenziali al sito di sintesi proteica.
c) r-RNA – ribosomiale.
I ribosomi sono organelli che svolgono funzioni di sintesi proteica.
5. Gli acidi nucleici differiscono nella localizzazione.
La quantità principale di DNA si trova nel nucleo della cellula (come parte dei cromosomi). Parte del DNA si trova nei mitocondri e nei cloroplasti (è chiamato DNA citoplasmatico). L'RNA si trova nel citoplasma.
4. Funzioni biochimiche di base dei nucleotidi
Pertanto, i nucleotidi uniscono un gruppo di sostanze che svolgono un'ampia varietà di funzioni:
1. Sono gli elementi costitutivi degli acidi nucleici e partecipano ai meccanismi molecolari mediante i quali l'informazione genetica viene immagazzinata, replicata e trascritta.
2. Svolgono un ruolo importante nel metabolismo energetico (fosforo), nell'accumulo e nel trasferimento di energia.
3. Servire come cofattori per enzimi appartenenti a varie classi.
4. Svolgono un ruolo importante nella sintesi e nella scomposizione di carboidrati, acidi grassi e lipidi.
5. Alcuni nucleotidi sono intermediari in complessi processi di trasduzione del segnale (trasmissione del segnale nelle cellule viventi).
Sezione 4. ENZIMI
Lezione 5. Struttura, meccanismo d'azione e classificazione degli enzimi
1. Struttura e proprietà fondamentali degli enzimi
Enzimi (enzimi)- sostanze di natura proteica che sono presenti in tutte le cellule viventi e agiscono come catalizzatori di processi biochimici.
In base alla loro composizione gli enzimi si dividono in:
1) semplice - costituito solo da aminoacidi;
2) complesso – composto da 2 parti:
Da una proteina chiamata apoenzima e
La parte non proteica è un cofattore.
Il complesso formato da un apoenzima e un cofattore è chiamato oloenzima.
Né l'apoenzima né il cofattore da soli sono in grado di catalizzare la reazione. Solo il loro complesso è funzionalmente attivo.
Tipi di cofattori:
Per la loro natura chimica, i cofattori possono essere rappresentati sia da composti organici che inorganici.
Cofattori organici possono essere divisi in due gruppi:
1) gruppi prostetici - cofattori che sono saldamente collegati all'apoenzima e, quando espulsi dal corpo, non sono disconnessi dalla parte proteica.
Ad esempio, il FAD fa parte dell'enzima succinato deidrogenasi del ciclo di Krebs.
2) coenzimi - cofattori che sono collegati agli apoenzimi da legami deboli e si separano facilmente da essi: ad esempio NAD, NADP e talvolta FAD.
Cofattori inorganici rappresentato da ioni metallici (il più delle volte ioni di ferro, rame, manganese, zinco, ecc.). Gli ioni metallici come cofattori partecipano direttamente all'atto della catalisi o formano ponti che collegano l'enzima al substrato.
Substrato (S)- una sostanza le cui trasformazioni chimiche sono catalizzate da un enzima.
La struttura dell'enzima, o enzima (E):

Poiché le molecole del substrato sono generalmente più piccole delle molecole degli enzimi, solo una parte della molecola dell'enzima, il centro attivo, entra in contatto diretto con il substrato. Inoltre, la forma geometrica della superficie di una porzione della molecola substrato è complementare alla superficie del centro attivo.
Il centro attivo di un enzima è una combinazione unica di residui di amminoacidi che garantisce l'interazione con la molecola del substrato e partecipa all'atto di catalisi. Negli enzimi complessi, il centro attivo include necessariamente un cofattore.
Il centro attivo può avere 2 sezioni:
· ancoraggio (substrato);
· catalitico.
La regione di ancoraggio ha somiglianza geometrica (corrispondenza) con la molecola del substrato e garantisce la specificità dell'azione enzimatica.
Somiglianze tra enzimi e catalizzatori non biologici
1. Qualsiasi catalizzatore (inorganico e organico) riduce l'energia di attivazione della molecola. L'energia di attivazione è la quantità di energia in calorie necessaria per convertire tutte le molecole di 1 mole di una sostanza in uno stato attivato, cioè uno stato in cui sono in grado di entrare in una reazione chimica.
2. Qualsiasi catalizzatore può accelerare solo le reazioni chimiche possibili dal punto di vista della termodinamica.
3. I catalizzatori non cambiano la direzione di una reazione chimica.
4. I catalizzatori non vengono consumati durante il processo di reazione.
Differenze tra enzimi e catalizzatori inorganici
1. La catalisi viene effettuata in condizioni molto blande (T, pH)
2. Alta efficienza: gli enzimi aumentano la velocità di reazione
10 10 - 10 12 volte.
Esempio: il corpo ha l'enzima catalasi (cofattore - Fe).
1 mg di ferro nella catalasi agisce come 10 tonnellate di ferro inorganico.
3. Specificità dell'azione. Ogni enzima accelera solo 1 reazione. Tipi di specificità:
Assoluto (1 enzima agisce solo su 1 substrato, ad esempio l'enzima ureasi catalizza l'idrolisi dell'urea);
Relativo (un enzima può agire su un gruppo di substrati strutturalmente simili).
4. La possibilità di una regolazione fine e precisa della velocità di reazione modificando le condizioni ambientali (legate alla natura proteica dell'enzima)
Ogni enzima ha la propria temperatura ottimale.
Esempio: temperatura corporea – 36,6 gradi; a T=40-41 gradi. Potrebbe verificarsi una denaturazione irreversibile. A basse temperature si osserva una diminuzione della velocità di catalisi enzimatica (a causa del movimento browniano delle molecole).
Gli enzimi sono molto sensibili ai cambiamenti dell'acidità dell'ambiente in cui operano. L'attività enzimatica avviene all'interno di una zona di pH abbastanza ristretta chiamata pH ottimale. Possiamo supporre che per ciascun enzima esista una certa concentrazione ottimale di protoni alla quale è più attivo.
Una variazione del pH porta ad una variazione delle cariche del centro attivo e della molecola nel suo insieme; di conseguenza, la conformazione della molecola proteica cambia, a seguito della quale la corrispondenza spaziale del centro attivo e del substrato viene interrotta, il che significa che la velocità di reazione diminuisce.
5. Possibilità di saturare l'enzima con il substrato (caratteristiche cinetiche).
6. La catalisi enzimatica è un processo strettamente programmato (1 reazione; 1 substrato; 1 enzima) - una serie di trasformazioni elementari di una sostanza, strettamente organizzate nello spazio e nel tempo.
2. Meccanismo d'azione degli enzimi
L'azione dell'enzima si basa sulla formazione di un complesso enzima-substrato. Sotto l'influenza del substrato, la conformazione dell'enzima cambia, quindi cambia il substrato.
Il meccanismo d’azione degli enzimi può essere rappresentato come il seguente diagramma:
E+S → ES → EZ → EP → E+P
Ci sono 4 fasi:
1. Si verificano giunzioni (ES) tra il substrato e l'enzima, in cui i composti sono collegati tramite legami ionici, covalenti o di altro tipo.
2. Sotto l'influenza dell'enzima attaccato, il substrato subisce modifiche (S→Z), rendendolo più accessibile per la reazione corrispondente.
3. Si verifica una reazione chimica per formare un complesso enzima-prodotto (EP).
4. I prodotti della reazione vengono rilasciati dal complesso enzima-prodotto.
3. Nomenclatura e classificazione degli enzimi
Nomenclatura degli enzimi (regole per la formazione dei loro nomi)
1. Casuale (basato su caratteristiche casuali) – banale
Esempio: papaina (carica papaja - dal legno).
2. Razionale: substrato + “aza” (lipidi – lipasi)
3. Sistematica: substrato + tipo di reazione catalizzata + “asi” (lattato deidrogenasi), oppure substrato + nome della classe a cui appartiene questo enzima + “asi” (lattato ossidoreduttasi).
Classificazione degli enzimi
Adottato nel 1961.
La classificazione si basa sul tipo di reazione catalizzata:
1. Ossidoreduttasi (enzimi complessi che catalizzano le reazioni redox). Esempio: isocitrato deidrogenasi del ciclo di Krebs.
2. Transferasi (catalizzano reazioni di trasferimento di gruppi funzionali o residui molecolari tra molecole). Esempio: chinasi – transferasi del 1° stadio della glicolisi.
3. Idrolasi (enzimi semplici che catalizzano le reazioni di idrolisi di amido, oligosaccaridi, grassi). Esempi: lipasi, invertasi, maltasi, ecc.
4. Liasi (catalizzano la scissione non idrolitica di alcuni gruppi di atomi da substrati con la formazione di un doppio legame o l'addizione a un doppio legame). Esempio: aldolasi dalla glicolisi.
5. Isomerasi (catalizzare reazioni di isomerizzazione - riarrangiamento spaziale o strutturale all'interno di una molecola). Esempio: triosefosfato isomerasi dalla glicolisi.
6. Ligasi (spesso chiamate sintetasi): catalizzano le reazioni di sintesi che comportano la rottura dei legami ricchi di energia (ATP).
Ogni enzima ha un codice di 4 cifre: classe-sottoclasse-sottoclasse-numero enzima individuale.
4. Cinetica delle reazioni enzimatiche
Una caratteristica della cinetica di una reazione enzimatica è la saturazione dell'enzima con il substrato, in cui un ulteriore aumento di [S] non porta ad un aumento della velocità di reazione. È stato empiricamente stabilito che la cinetica di una reazione enzimatica può essere espressa dal seguente grafico:

La concentrazione del substrato alla quale l'enzima raggiunge la saturazione è una caratteristica costante per ciascun enzima specifico.
La cinetica di una reazione enzimatica può essere descritta utilizzando un'equazione derivata teoricamente dagli scienziati Michaelis e Menten, da cui prende il nome.
Equazione di Michaelis-Menten
Km – Costante di Michaelis. Questa è la concentrazione del substrato alla quale la velocità di reazione è la metà del massimo.
La costante di Michaelis caratterizza l'affinità dell'enzima per il substrato: minore è questa costante, maggiore è l'affinità dell'enzima per il substrato, più efficiente è la reazione.
5. Regolazione dei processi enzimatici nella cellula
Numerosi metodi per la regolazione dei processi enzimatici possono essere suddivisi in due gruppi:
1. Regolazione del contenuto dell'enzima modificando la velocità della sua sintesi e degradazione. È necessario notare i seguenti processi:
la repressione è il processo di soppressione (o riduzione) della velocità di sintesi enzimatica;
l'induzione è il processo di accelerazione della sintesi di enzimi sotto l'influenza di specifici composti a basso peso molecolare - induttori.
2. Regolazione dell'attività degli enzimi presenti nella cellula.
a) modificando la temperatura, il valore del pH, la quantità di substrato, cofattori, ecc.;
b) regolazione allosterica (caratteristica solo degli enzimi allosterici). Gli allosterici sono enzimi che hanno, oltre al centro attivo, un ulteriore centro legante (centro allosterico). L'attività degli enzimi allosterici è regolata da un cambiamento nella conformazione delle molecole enzimatiche causato dall'aggiunta di uno speciale metabolita al centro allosterico. Il regolatore del metabolita (effettore allosterico) funziona sia come attivatore che come inibitore;
c) modifica covalente degli enzimi - la regolazione dell'attività catalitica degli enzimi può essere effettuata mediante l'aggiunta covalente di un gruppo fosfato o nucleotide. Ad esempio, la forma fosforilata della glicogeno fosforilasi ha un'attività catalitica più elevata;
d) modifica dell'attività degli enzimi con l'aiuto di attivatori - composti chimici che aumentano l'attività degli enzimi (ad esempio, l'amminoacido cisteina e il tripeptide glutatione attivano l'azione di molte proteasi).
e) modifica dell'attività degli enzimi mediante inibitori: composti chimici che sopprimono l'attività degli enzimi.
Inibizione
Inibizione– riduzione o soppressione completa dell’attività enzimatica sotto l’influenza di determinate sostanze (inibitori).
L'inibizione può essere di due tipi principali: irreversibile e reversibile.
A irreversibile Quando inibiti, l'enzima e l'inibitore formano un complesso non dissociante. L'inibizione irreversibile nel corpo è rara e quando si verifica è dovuta a sostanze provenienti dall'esterno.
A reversibile Quando inibiti, l'enzima e l'inibitore formano un complesso dissociante.
L'inibizione reversibile, a sua volta, è divisa in competitiva e non competitiva.
Inibizione competitiva– inibizione, in cui il substrato e l'inibitore hanno una struttura simile e competono per il sito attivo dell'enzima. L'inibizione competitiva è comune nel corpo ed è un modo per regolare l'attività enzimatica.
La velocità di reazione durante l'inibizione competitiva dipende dal rapporto tra le concentrazioni di substrato e inibitore. Maggiore è la concentrazione del substrato, maggiore è la probabilità di formazione di complessi, maggiore è la velocità di reazione. Pertanto, l’inibizione competitiva può essere soppressa aumentando la concentrazione del substrato.
Inibizione non competitiva– inibizione, in cui il substrato e l'inibitore interagiscono con diverse parti della molecola enzimatica. In questo caso l'inibitore, combinandosi con la molecola dell'enzima, ne modifica la struttura in modo tale che è impossibile raggiungere la massima velocità di reazione.
Nell'inibizione non competitiva, l'aumento della concentrazione del substrato non elimina l'effetto dell'inibitore. L'inibizione non competitiva nel corpo è solitamente associata all'ingresso di metalli pesanti nel corpo.
Sezione 5. CARBOIDRATI E LORO METABOLISMO
Lezione 6. Struttura chimica e proprietà dei carboidrati
1. Caratteristiche generali e classificazione dei carboidrati
I carboidrati includono composti che hanno proprietà diverse e spesso completamente opposte. Tra questi ci sono sostanze a basso e alto peso molecolare, cristalline e amorfe, altamente solubili in acqua e completamente insolubili in essa, capaci di ossidazione e relativamente resistenti all'azione degli agenti ossidanti.
La formula generale caratteristica della stragrande maggioranza dei carboidrati è C n (H 2 O) m
In base alla loro natura chimica i carboidrati si dividono in:
· monosaccaridi (zuccheri semplici);
· oligosaccaridi;
· polisaccaridi.
I monosaccaridi contengono 3-8 atomi di carbonio e non subiscono idrolisi per formare idrocarburi semplici.
Gli oligosaccaridi sono polimeri di monosaccaridi che contengono 2-10 residui di monosaccaridi.
I polisaccaridi sono polimeri di monosaccaridi che contengono più di 10 residui monosaccaridi.
2. Struttura, proprietà e funzioni dei monosaccaridi
Monosaccaridi sono suddivisi nei seguenti gruppi:
1. In base al numero di atomi di carbonio:
· Triosi (3)
Tetrosi (4)
· Pentosi (5)
Esosi (6)
Eptosio (7)
Poltoso (8)
2. Per struttura chimica:
· Aldoso
Tutti i monosaccaridi sono alcoli, alcoli aldeidici o chetoalcoli. Nelle loro molecole, di regola, il numero di atomi di carbonio è uguale al numero di molecole d'acqua (cioè m = n).
D-glucosio (aldosio) D-fruttosio (chetosio)
Aldosi e chetosi sono isomeri.
Proprietà chimiche di base dei monosaccaridi:
1. La mutarotazione è la transizione di un anomero da una forma all'altra (ad esempio, α-glucosio → β-glucosio). Gli anomeri sono forme enantiomeriche di monosaccaridi che differiscono nella posizione dell'idrossile emiacetale.
2. Riduzione ad alcoli polivalenti (ad esempio, il glucosio viene ridotto a sorbitolo, il ribosio a ribitolo).
3. Ossidazione con formazione di acidi corrispondenti (ad esempio, a seconda del gruppo ossidato, il glucosio può formare acidi gluconico, glucuronico e glucarico).
4. Epimerizzazione (ad esempio, in un ambiente leggermente alcalino, il D-glucosio è in equilibrio con chetoesoso (D-fruttosio) e aldoesoso (D-mannosio).
5. Formazione di glicosidi. La condensazione del gruppo OH anomerico con il gruppo alcolico della molecola porta alla formazione di O-glicosidi. È attraverso questi legami che si formano oligo e polisaccaridi. Quando un gruppo OH anomerico interagisce con un gruppo NH 2, si formano N-glicosidi.
6. Esterificazione. I gruppi idrossilici dei monosaccaridi formano esteri con vari acidi. La fosforilazione degli zuccheri svolge un ruolo particolarmente importante nel metabolismo.
7. La capacità di reagire con composti contenenti azoto ad alte temperature con la formazione di sostanze colorate specifiche: le melanoidine.
8. La capacità del glucosio (e degli altri esosi) di essere scomposto (mediante glicolisi) e fermentato dai microrganismi.
Principali funzioni dei monosaccaridi:
1. Energia (i monosaccaridi vengono facilmente scomposti, rilasciando energia che viene spesa nella formazione di ATP).
2. Plastica (metabolica). I monosaccaridi sono precursori per la formazione di molte sostanze importanti: polisaccaridi di riserva e strutturali, aminoacidi, acidi grassi, glicerolo, ecc.
3. Struttura, proprietà e funzioni degli oligosaccaridi
Gli oligosaccaridi differiscono per le seguenti caratteristiche:
1. La quantità di monosaccaridi.
2. Composizione di alta qualità.
3. La natura del legame glicosidico tra monosaccaridi.
Nelle soluzioni i monosaccaridi sono sempre presenti in forma ciclica; Sono anche inclusi negli oligo e polisaccaridi in forma ciclica.
Il primo atomo di carbonio collegato all'ossigeno è il più reattivo. Di norma, il legame si forma a causa dell'idrossile glicosidico (emiacetale).
Gli oligosaccaridi sono caratterizzati da alcune proprietà note per i monosaccaridi. Va anche notato che gli oligosaccaridi che entrano nel corpo umano con il cibo subiscono idrolisi nel tratto gastrointestinale nei loro blocchi strutturali: i monosaccaridi. Pertanto, entrano nelle cellule sotto forma di zuccheri semplici e, di conseguenza, svolgono le stesse funzioni dei monosaccaridi.
Tra gli oligosaccaridi i più diffusi sono i disaccaridi. Consideriamo la composizione chimica dei più importanti.
Saccarosioè costituito da residui di α-glucosio e β-fruttosio collegati da un legame β-glicosidico (o fruttosidico). L'idrolisi del saccarosio avviene con la partecipazione dell'enzima invertasi (saccarasi):

saccarosio α-glucosio β-fruttosio
L'invertasi si trova in grandi quantità nel lievito e nell'intestino degli organismi. Una miscela di glucosio e fruttosio in uguali quantità, che si forma durante l'idrolisi del saccarosio, è chiamata zucchero invertito.
Maltosio– un disaccaride costituito da 2 residui di α-glucosio. È il prodotto principale dell'idrolisi dell'amido.
Maltosio → α-glucosio + α-glucosio
L'idrolisi del maltosio avviene con la partecipazione dell'enzima maltasi.
La maltasi si trova nella saliva e nel succo pancreatico.
Il lattosio è lo zucchero del latte prodotto nel corpo degli animali.
Lattosio= β-galattosio + α-glucosio L'idrolisi del lattosio è catalizzata dall'enzima lattasi.
La lattasi è molto attiva nei neonati; Alcuni adulti non trattengono la lattasi, il che si traduce in un’intolleranza al latte.
4. Struttura, proprietà e funzioni dei polisaccaridi
I polisaccaridi si dividono in omosaccaridi ed eterosaccaridi.
Parte omosaccaridi comprende monosaccaridi dello stesso tipo. Se il monomero è fruttosio, allora il polisaccaride si chiama fruttano; galattosio – galattano; il glucosio è un glucano.
Monomeri eteropolisaccaridi sono monosaccaridi di 2 o più tipi. Ad esempio, l'arabinosio e il glucosio fanno parte degli arabinoglucani; arabinosio e xilosio sono arabinoxilani.
Amido (omosaccaride)– polisaccaride di riserva delle piante; esiste in 2 forme: amilosio e amilopectina.
Amilosio– polisaccaride lineare, costituito da residui α-glucosio collegati da legami α–1,4.
Amilopectina– un polisaccaride ramificato, in cui per ogni 12 residui di glucosio collegati da un legame α–1,4 esiste un legame α–1,6.
Queste sostanze variano notevolmente nelle loro proprietà fisiche e chimiche. Ad esempio, lo iodio colora l'amilosio blu e l'amilopectina rosso-viola. Differiscono anche nella solubilità: l'amilosio si dissolve facilmente in acqua calda e dà soluzioni con una viscosità relativamente bassa, mentre l'amilopectina si dissolve in acqua solo se riscaldata sotto pressione e dà soluzioni molto viscose.
Glicogeno(“amido animale”) – simile come struttura all’amido, ma caratterizzato da una maggiore ramificazione.
È un nutriente di riserva (formato principalmente nel fegato e nei muscoli).
La cellulosa (fibra) è un polisaccaride costituito da un gran numero di residui di β-glucopiranosio.
Funzioni dei polisaccaridi:
1. Fornitura di nutrienti (amido, glicogeno - le sostanze più comuni).
2. Fonti energetiche (se utilizzate come fonti energetiche, devono prima essere scomposte in monosaccaridi).
3. Strutturale (la cellulosa forma le pareti cellulari nelle piante, la chitina negli animali, la mureina nei batteri).
Abstract sull'argomento:
Separazione delle dimensioni delle proteine utilizzando DDC-Na
Separazione delle dimensioni delle proteine utilizzando DDC-Na
L'elettroforesi delle proteine in un sistema semplice è utile per la separazione delle proteine, ma non per la caratterizzazione. La mobilità elettroforetica di ciascuna proteina in un sistema semplice dipende simultaneamente dalla massa della proteina, dalla sua carica elettrica totale, dalla configurazione e dalla rigidità dell'impaccamento del globulo proteico. Il contributo di ciascuno di questi fattori non è noto e può variare in modo significativo a seconda delle condizioni dell'elettroforesi. Per stabilire una stretta correlazione tra uno qualsiasi dei parametri elencati e la mobilità elettroforetica della proteina, è necessario escludere l'influenza di tutti gli altri.
L'elettroforesi PAGE utilizzando DDC-Na consente la separazione di proteine che differiscono tra loro solo per il peso molecolare. Per fare ciò, la miscela proteica nella preparazione originale viene trattata con un eccesso di DDC-Na non inferiore a tre volte (in peso). A causa delle interazioni idrofobiche, il detergente si lega equamente alla stragrande maggioranza delle proteine in un rapporto di 1,4 mg di DDC-Na per 1 mg di proteine.
L'enorme eccesso di residui di acido solfonico completamente dissociati introdotti dal detersivo rende nella maggior parte dei casi irrilevante il ruolo della carica propria delle proteine. A causa della repulsione elettrostatica dei residui ravvicinati di acido solforico caricati negativamente, la catena proteica si raddrizza e assume la forma di un'asta rigida con un diametro di -1,6 m e una lunghezza che dipende solo dal numero di anelli di questa catena, e quindi sul peso molecolare della proteina.
Contemporaneamente all'elaborazione del DDC-Na, è necessario garantire la possibilità del completo dispiegamento della catena proteica e, a questo scopo, rompere tutti i legami covalenti S-S all'interno della molecola proteica. A questo scopo, prima dell'elettroforesi, la proteina viene trattata anche con un'alta concentrazione (1%) (3-mercaptoetanolo a temperatura elevata.
La mobilità elettrofretica, cioè la velocità di migrazione ad un'intensità di campo di 1 V/cm, del complesso proteico rigido DDC-Na risulta essere correlata al peso molecolare della proteina (M) dal semplice rapporto A-BIgM, dove A e B sono coefficienti dipendenti dalla porosità del gel, dalla temperatura e da altre condizioni sperimentali. È più conveniente rappresentare il valore "e" in unità relative, esprimendo il rapporto tra i percorsi di migrazione della proteina e il "colorante principale" - blu di bromofenolo. Indichiamo questo rapporto Rf. Tale sostituzione influenzerà solo il valore dei coefficienti A e B, di cui sappiamo solo che sono nei dati, le condizioni sperimentali sono le stesse per tutte le proteine. Anche i valori modificati dei coefficienti saranno valori costanti in questo esperimento, invece di determinare loro, utilizzano il metodo del confronto con “marcatori” conosciuti dalla loro massa Contemporaneamente all'elettroforesi della miscela di proteine in studio, una traccia separata sulla stessa piastra, cioè, in identiche condizioni sperimentali, una miscela di marcatori noti. è separato. Con il loro aiuto, la dipendenza logM = f (Rf) è costruita da punti, che, naturalmente, risulta essere lineare. Sulla base di questa dipendenza, è possibile determinare graficamente i valori di log M da valori misurati di Rf. e quindi M per la proteina A in studio Naturalmente, tutti i pretrattamenti con detergente e P-mercaptoetanolo dovrebbero essere eseguiti esattamente allo stesso modo per questa proteina e per tutti i marcatori.
La scelta del tampone in questa variante non ha alcun ruolo, poiché la carica della proteina è determinata dal suo complesso con DDC-Na. In genere, viene utilizzato un tampone neutro con l’aggiunta di DDC-Na allo 0,1% per mantenere il complesso detergente-proteina.
Per le proteine con peso molecolare inferiore a 12mila dalton, la determinazione di M diventa inaffidabile. Per diversi intervalli di massa proteica, si consiglia di utilizzare PAGE di diverse porosità, secondo la seguente tabella:
Intervallo M (migliaia di dalton) % PAAG 12-43 15 16-68 10 36-94 7,5 57-212 5
Il processo di elettroforesi e colorazione delle proteine dopo il completamento viene eseguito come di consueto, ma è preferibile lavare il DDC-Na dalle proteine prima della colorazione immergendo il gel in TCA al 50% per una notte.
Il DDC-Na in complesso con le proteine previene in una certa misura la colorazione (grande carica negativa!).
Elettroforesi bidimensionale della PAGINA
La separazione completa di una miscela complessa di proteine non può sempre essere ottenuta in un esperimento elettroforetico. Esiste sempre la possibilità che in un dato sistema elettroforetico diverse proteine migrino nella stessa zona, sia a causa della vicinanza delle loro dimensioni, sia per la coincidenza delle loro mobilità elettroforetiche ad un valore di pH selezionato, o, infine, come una risultato di una combinazione di questi parametri sfavorevole alla separazione. Pertanto, in casi complessi di frazionamento di una miscela di proteine, ha senso utilizzare la separazione nella prima direzione come iniziale per la separazione nella seconda, perpendicolare alla prima direzione in condizioni di elettroforesi modificate.
Per fare ciò, la traccia della prima direzione (senza sedimentazione e colorazione delle proteine al suo interno) viene ritagliata e posizionata sulla zona iniziale della piastra della seconda direzione (senza tasche). Il contatto tra due gel è assicurato riempiendo il punto del loro contatto con una soluzione di agarosio fusa nello stesso tampone. L'elettroforesi, naturalmente, viene eseguita in direzione perpendicolare alla striscia. Ciascuna banda disomogenea in essa contenuta può dare diversi spot nella seconda direzione se, in nuove condizioni, le proteine in essa contenute acquisiscono mobilità elettroforetiche diverse. Di conseguenza, dopo la deposizione e la colorazione, sulla lastra appare un motivo di macchie distribuite su tutta la superficie (“impronta digitale”). Il numero di macchie di diverse proteine che possono essere registrate su una piastra può raggiungere diverse centinaia. Nella fig. 42 riproduce lo schema della distribuzione delle macchie ottenuto mediante elettroforesi bidimensionale delle proteine della subunità grande di uno dei batteri (nel lavoro di Mets, Bogorad Anal. Biochem. 57 200, 1974).
Estrazione delle proteine dal gel dopo elettroforesi
Per scopi di identificazione analitica, le proteine della PAGE (senza precipitazione o colorazione) possono essere trasferite su un filtro a membrana di nitrocellulosa. Tali filtri hanno la capacità di assorbire le proteine basiche. Il trasferimento viene effettuato lavando via le bande proteiche dal gel con una corrente tampone in una direzione perpendicolare alla superficie della piastra. Il filtro viene applicato direttamente sul gel bagnato. L'apparecchio per fornire il flusso tampone dal gel al filtro verrà descritto di seguito in relazione all'elettroforesi del DNA.
Di conseguenza, il filtro produce “repliche” delle proteine separate nel gel. Per identificarli si possono utilizzare reazioni caratteristiche, talvolta enzimatiche o immunitarie (vedi sotto), nonché l'ibridazione con DNA o RNA marcati con fosforo radioattivo, se queste proteine legate in vivo al DNA o proteine ribosomiali che si legano all'RNA ribosomiale sono state sottoposte a separazione.
La cosa principale che, dal punto di vista dell'elettroforesi, distingue gli acidi nucleici dalle proteine è la significativa carica negativa totale causata dalla dissociazione di numerosi residui di acido fosforico nei legami tra nucleosidi. Il pH dell'ambiente ha poco effetto su questa carica. Pertanto, l'elettroforesi può essere eseguita non in un tampone, ma in qualsiasi soluzione contenente ioni adatta, ad esempio in una soluzione alcalina debole. In tutti i casi, l'EDTA viene aggiunto al mezzo liquido ad una concentrazione di 1-2 mM. Ciò è necessario per bloccare l'azione delle nucleasi e impedire la precipitazione (soprattutto dell'RNA) da parte dei metalli bivalenti.
Pertanto, la separazione dei frammenti di DNA e RNA mediante elettroforesi deve essere effettuata solo in base alla dimensione. Tuttavia, queste dimensioni possono variare in un intervallo molto ampio: da decine di unità nucleotidiche a molte centinaia di migliaia e, se espresse in termini di pesi molecolari, da diverse migliaia a centinaia di milioni di dalton.
Naturalmente, per il frazionamento di frammenti relativamente corti, viene utilizzata l'elettroforesi PAGE e per la separazione del DNA ad alto peso molecolare viene utilizzato un vettore a pori più grossolani: l'agarosio. La conosceremo un po' più tardi. Nel frattempo, in relazione ai paragrafi precedenti, è opportuno fare alcuni commenti sull'elettroforesi del DNA in PAGE. La tabella seguente fornisce raccomandazioni per la scelta della porosità del gel in base alla dimensione dei frammenti di DNA relativamente corti, caratterizzati dal numero di coppie di basi (bp):
|
Gamma P.O (cose) |
||||||
L'ultimo intervallo di questa tabella contiene le dimensioni massime del DNA disponibili per il sequenziamento automatico dei nucleotidi. Questo metodo rivoluzionario di analisi del DNA sarà discusso nel prossimo capitolo.
Ora, prima di passare all'elettroforesi dell'agarosio, desidero presentare a studenti e lettori nuove idee per frazionare grandi frammenti di DNA in PAGE utilizzando l'uso originale di impulsi di tensione elettrica applicati al gel. Questi impulsi vengono applicati alternativamente in due direzioni reciprocamente perpendicolari. L'idea qui è questa. Anche una molecola di DNA molto lunga e flessibile può passare attraverso i pori relativamente piccoli del gel se viene estesa nella direzione del movimento verso l'anodo “principale”, ad esempio situato sul fondo della piastra. La tensione viene fornita a questo anodo non costantemente, ma a impulsi. Il secondo anodo “ausiliario” crea un’intensità del campo elettrico perpendicolare alla direzione principale del movimento del DNA verso il fondo della piastra. La tensione gli viene fornita anche con impulsi abbastanza potenti, ma più brevi rispetto all'anodo principale, alternati ad essi.
Lo scopo del campo elettrico "perpendicolare" è quello di ruotare le lunghe molecole di DNA di grandi dimensioni in modo che, nel momento in cui viene applicato l'impulso "longitudinale", siano in una posizione favorevole allo spostamento lungo la piastra verso l'anodo principale. Più lunghe sono le catene del DNA, più lentamente cambieranno il loro orientamento e quindi più lentamente si muoveranno nella direzione desiderata. Gli autori del metodo affermano che in questo modo sono riusciti a separare frammenti di DNA fino a cinque milioni coppie di basi.
Gel di agarosio
L'agarosio è una frazione particolarmente pura di un polisaccaride lineare naturale, l'agar, isolato dalle alghe marine. Ci siamo già incontrati.
Il peso molecolare dei singoli filamenti di agarosio è compreso tra 10 e 100 mila dalton. L'agarosio per l'elettroforesi viene fornito sotto forma di polvere liofilizzata (essiccata sotto vuoto). La gelificazione avviene quando anche una soluzione calda molto diluita di agarosio in un tampone si raffredda. Ad una temperatura di 84-96°C (e per alcuni tipi di agarosio già a 70°), i filamenti polimerici si sciolgono e formano un liquido trasparente omogeneo con l'ambiente acquoso circostante. Ha un'isteresi di temperatura pronunciata: congela a una temperatura di circa 40°C. Una volta raffreddate a questa temperatura, anche le soluzioni di agarosio allo 0,4% formano gel forti. Per i tipi di agarosio a basso punto di fusione, la temperatura di solidificazione è ridotta a 30°. Questa caratteristica dell'agarosio facilita tutte le manipolazioni con le sue soluzioni senza timore che si induriscano. Inoltre, la sospensione di agarosio sciolta in un bagno bollente in acqua viene raffreddata a 50-55° e solo a questa temperatura si aggiunge il tampone concentrato e tutti gli altri additivi, per poi versare in uno stampo per l'elettroforesi. Ciò è conveniente e non è associato al verificarsi di deformazioni termiche.
Durante il raffreddamento, i filamenti orientati in modo casuale di agarosio solidificato, grazie a molteplici legami idrogeno tra i filamenti, si assemblano in fasci. Questi fili, intersecandosi liberamente, creano nel liquido che li circonda una rete spaziale molto ampia, porosa e allo stesso tempo rigida.
La dimensione dei pori di un gel di agarosio è naturalmente correlata alla concentrazione della sua soluzione originale. Si possono dare alcune raccomandazioni per la scelta di questa concentrazione, tratte dall'esperienza dell'elettroforesi degli acidi nucleici:
Per gli acidi nucleici di virus e grandi plasmidi 0,4-0,5%.
Per i restrittori del DNA contenenti 5-20 mila paia di basi, 0,7-0,8%.
Per le restrizioni sui reovirus a DNA e RNA a doppio filamento più corti, 1,5%.
Per l'RNA ribosomiale - 1,75%.
Per restrizioni di mRNA e DNA fino a 1000 bp. -2%
Preparare una piastra per elettroforesi di agarosio è molto semplice. Il vetro della dimensione richiesta è ricoperto su tutti i lati lungo il bordo con nastro adesivo continuo in modo che il suo bordo superiore sporga di alcuni millimetri sopra la superficie del vetro. Quest'ultimo è posto su un tavolo rigorosamente orizzontale. Il volume calcolato di soluzione di agarosio ancora liquida viene versato direttamente sul vetro. Quindi viene installato un pettine simile a quello che viene posizionato nello stampo PAGE prima della sua polimerizzazione vicino a un bordo del vetro. Solo che questa volta il pettine viene immerso nell'agarosio perpendicolarmente al vetro (i suoi denti, però, non devono toccare il vetro). Dopo che l'agarosio si è indurito, il pettine viene rimosso, formando così dei “pozzetti” per i preparati. L'elettroforesi viene effettuata in posizione orizzontale (in tracce), applicando stoppini provenienti da serbatoi con tamponi. Il valore dell'intensità del campo operativo è 2-5 V/cm.
Il DNA, soprattutto quelli a doppio filamento, così come l'RNA, sono ben colorati con un colorante fluorescente giallo - bromuro di etidio. Questo colorante trasporta una carica positiva, che gli consente di interagire con i gruppi fosfato degli acidi nucleici. Inoltre, è in grado di intercalare (incorporare) tra le basi del DNA a doppio filamento, il che porta ad un forte aumento della sua fluorescenza quando illuminato con luce ultravioletta. La colorazione può essere eseguita anche dopo l'elettroforesi immergendo il gel per 0,5-1 ora in una soluzione acquosa del colorante (1 μg/ml) o iniettandolo direttamente nel gel. In quest'ultimo caso è possibile monitorare il movimento delle bande nel gel. In questo caso la lastra di vetro viene illuminata dal basso con una lampada UV. La sensibilità al colore è elevata. È possibile osservare e fotografare bande contenenti 0,01 µgDNA.
Al termine dell'elettroforesi, il DNA può essere fissato nel gel mediante precipitazione dalla soluzione immergendo il gel in etanolo al 70%.
Pipette
Naturalmente, le micropipette moderne non sono affatto simili a quelle utilizzate nel laboratorio di chimica della scuola. Questa è la loro struttura. L'impugnatura (esterna) in plastica, piuttosto voluminosa, della pipetta può essere comodamente tenuta con quattro dita, lasciando il pollice libero di premere il pulsante che sporge dall'estremità superiore dell'impugnatura. Dal basso emerge una lunga asta metallica leggermente conica, all'estremità della quale è fissata saldamente una punta conica cava in plastica speciale.
La maniglia contiene un meccanismo, la cui parte principale è costituita da un cilindro sottile montato con precisione e da un pistone, premuto verso l'alto da una molla. Premendo il pulsante si sposta il pistone verso il basso. Abbassando la punta nel liquido e rilasciando il pulsante precedentemente premuto, aspirare un volume di 1, 2, 3 o più microlitri nella punta, in base alla calibrazione della pipetta. Premendo una seconda volta il pulsante, il liquido viene espulso completamente dalla punta. Le punte vengono fornite sterilizzate e sono monouso.
Esistono pipette con la possibilità di preregolare il volume del liquido, ad esempio da 2 a 20 microlitri. La regolazione viene effettuata ruotando la testa della vite, che imposta la lunghezza della corsa del pistone. Il volume selezionato è indicato da un tamburo graduato associato alla vite.
Effetto della struttura secondaria del DNA
Nel comportamento elettroforetico delle molecole di acido nucleico a filamento singolo (DNA denaturato, RNA) e a doppio filamento, molto è determinato dalla loro dimensione. Nel caso delle catene polinucleotidiche corte, la loro molecola nativa a doppio filamento ha una struttura più rigida rispetto a una molecola a filamento singolo della stessa dimensione. Si piega più difficilmente quando attraversa la rete spaziale del gel. Per questo motivo, frammenti di DNA a doppio filamento relativamente corti rimarranno indietro rispetto al DNA denaturato della stessa lunghezza durante l'elettroforesi PAGE. Questa situazione si verificherà anche per il DNA del batteriofago FH-174 con un peso molecolare di 3,5 milioni di dalton. Tuttavia, per molecole più grandi la situazione può essere invertita. La lunga catena a doppio filamento risulta essere piuttosto flessibile: si muove attraverso i pori del gel come se “dimenasse come un serpente”. Nel frattempo, una catena a filo singolo della stessa lunghezza si piega in una “palla caotica” allentata di dimensioni tali che il suo movimento nel gel è più difficile. Il DNA denaturato in questo caso resta indietro rispetto al DNA nativo durante l'elettroforesi. Naturalmente il limite di inversione dell'effetto descritto dipende dalla dimensione dei pori del gel.
Il DNA virale e mitocondriale a doppio filamento, così come i plasmidi batterici, hanno la struttura di un anello chiuso a doppio filamento. Lo stato nativo di un tale anello è “super-ritorto” (corrisponde ad un minimo di tensioni interne). L'anello nel suo insieme è piegato a “fascio”, il che ne aumenta notevolmente la compattezza (forma I). Se si verifica una singola rottura della catena zucchero-fosfato in almeno uno dei due fili ritorti dell'anello, il fascio si apre e, sotto l'influenza delle forze di repulsione elettrostatica dei gruppi fosfato, l'anello si raddrizza. La compattezza della molecola diminuisce, le sue dimensioni esterne aumentano (forma II). Per quanto riguarda una molecola di DNA lineare a doppio filamento (forma III), a seconda della dimensione media dei pori del gel, può migrare più velocemente o più lentamente di un anello superavvolto della stessa massa. Per un gel a grande porosità, la compattezza della Forma I può essere un fattore decisivo; per i pori più piccoli viene in primo piano la maggiore flessibilità della molecola di DNA lineare (forma III).
La velocità di migrazione delle molecole di DNA lineare a doppio filamento diminuisce con l'aumentare della loro massa, ma solo fino a un certo limite. Con un peso molecolare superiore a 5 milioni in un gel di agarosio all'1,6% e ad un peso molecolare superiore a 12 milioni di dalton in un agarosio allo 0,8%, le molecole migrano essenzialmente alla stessa velocità, indipendentemente dalla loro massa. In questi casi, la flessibilità gioca un ruolo decisivo: la capacità di molecole molto lunghe, quando si attorcigliano, di passare attraverso il gel con la stessa facilità (o con uguale difficoltà) a qualsiasi lunghezza.
L'RNA ribosomiale può avere una struttura secondaria che è significativamente più sfavorevole alla migrazione in un gel rispetto al DNA. Il fatto è che nelle grandi molecole di RNA questa struttura è rappresentata da numerose "forcine" che sporgono in tutte le direzioni. Questi sono luoghi di accoppiamento locale in strutture rigide a doppio filamento di individui, spesso molto distanziati tra loro lungo la sequenza principale, regioni di RNA complementari. Una tale molecola non può più muoversi attraverso i pori del gel. Quando si assembla il “sandwich” principale (gel, filtro e fogli di carta da filtro che li inseriscono), è necessario verificare che non rimangano bolle d'aria tra di essi.
Il trasferimento del DNA su un filtro di nitrocellulosa richiede 2-3 ore. Va notato che i frammenti corti di DNA vengono trattenuti male su un filtro di nitrocellulosa. Se questo è significativo, è meglio utilizzare filtri realizzati con la cosiddetta "carta diazo" o "carta DBM". La società Whatman lo produce con il nome “Whatman 540”.
Letteratura
1 Kurashvili L.V. Disturbi del metabolismo lipidico negli stati di tensione. II Letture di Zakharyinsky. Estratti delle relazioni. - 1995. -P.127.
2 Kurashvili L.V. Attività della lipasi e LCAT durante la disidratazione a lungo termine. Nel libro: Problemi attuali nella diagnosi, nel trattamento e nella riabilitazione dei pazienti. Estratti delle relazioni. - Penza, 1995. -P.71-72.
3 Kurashvili L.V. Stato fosfolipidico in caso di alterato metabolismo idroelettrolitico. Nel libro: Problemi attuali nella diagnosi, nel trattamento e nella riabilitazione dei pazienti. Estratti delle relazioni. - Penza, 1995.-P.69-70.
La dialisi è il processo di separazione delle sostanze ad alto peso molecolare da quelle a basso peso molecolare utilizzando membrane semipermeabili. Le molecole proteiche, avendo un grande peso molecolare, non sono in grado di penetrare attraverso le partizioni semipermeabili (membrane artificiali e naturali). In questo caso, le particelle a basso peso molecolare (organiche e inorganiche) passano facilmente attraverso i pori delle membrane semipermeabili.
La dialisi è ampiamente utilizzata per purificare le proteine dalle impurità a basso peso molecolare (sale, zucchero e altre), che passano facilmente attraverso i pori delle membrane semipermeabili. Il dispositivo in cui viene eseguita la dialisi è chiamato dializzatore. Un sacchetto di cellophane o collodio posto in un recipiente con acqua rappresenta il dializzatore più semplice (Fig. 1). La proteina inserita nel sacchetto rimane al suo interno e le sostanze a basso peso molecolare si diffondono nell'acqua attraverso la membrana.
L’albumina e le globuline possono essere separate mediante dialisi. Quando i sali passano da una soluzione proteica nell'ambiente, le globuline precipiteranno, perché sono insolubili in un ambiente acquoso e le albumine rimarranno in soluzione. Il dializzatore più semplice può essere un sacchetto di plastica posto in un bicchiere d'acqua. Una soluzione salina della proteina viene posta in un sacchetto, mentre le molecole di sostanze a basso peso molecolare (ioni di sale) si diffondono attraverso la parete del sacchetto e le grandi molecole proteiche rimangono all'interno del sacchetto.
2 NÍ 4 + SO 4 2-
Figura 1 - Schema del dializzatore
Progresso. 10-15 ml di una soluzione di albume contenente albumine, globuline e solfato di ammonio vengono posti in un sacchetto di plastica e immersi in un bicchiere di acqua distillata in modo che il livello del liquido nella busta corrisponda al livello dell'acqua. Per accelerare la dialisi è necessario cambiare l'acqua nel vaso. Dopo 1 ora si analizza l'acqua del bicchiere: in una provetta si effettua una reazione con il biureto e ci si assicura che le proteine non passino attraverso la membrana; in un'altra provetta si effettua una reazione per lo ione SO 4 2- aggiungendo alcune gocce di una soluzione di BaCI 2 al 10% per stabilire la penetrazione del sale attraverso la parete del sacchetto di plastica. Dopo 2-3 ore all'interno del sacchetto appare un precipitato di globulina che viene filtrato. Le proteine della frazione albuminica rimangono nel filtrato.
La natura globulina delle proteine del precipitato viene dimostrata sciogliendola in soluzioni saline (soluzione al 10% di cloruro di ammonio, cloruro di sodio, ecc.). Quando la soluzione viene diluita con acqua, si forma nuovamente un precipitato.
La natura albuminica delle proteine filtrate è confermata dalla diluizione con acqua in assenza di sedimenti o dalla completa saturazione con solfato di ammonio (forme precipitate).
Materiali e reagenti: una soluzione di albume contenente albumine, globuline e solfato di ammonio (vedi Appendice B, paragrafo 5); Soluzione di cloruro di bario al 10%; Soluzione di idrossido di sodio al 10%; Soluzione all'1% di solfato di rame; Soluzione di cloruro di ammonio al 10%; solfato di ammonio, macinato finemente.
Attrezzatura: provette ordinarie; sacchetto di cellophane o collodio; bicchiere o recipiente per 1–2 ml; Pipette dosatrici da 10 ml; pipette.
Domande di controllo
1. Il principio del processo di dialisi.
2. Il dispositivo di un semplice dializzatore.
3. Per quali scopi viene utilizzata la dialisi?
4. Perché le globuline (proteine sali solubili) possono essere separate dalle globuline (proteine idrosolubili) mediante dialisi?
5. Come dimostrare la natura globulina e albumina delle proteine durante la loro separazione?
Separazione delle proteine (frazionamento).
Spesso utilizzato per separare le proteine metodi cromatografici , basato sulla distribuzione delle sostanze tra due fasi, di cui una mobile e l'altra immobile. Nella cromatografia liquida, la zona delle sostanze separate, con l'aiuto di una corrente di liquido eluente (lavaggio), si sposta rispetto alla fase stazionaria, che ha affinità diverse per i componenti da separare. Quando la zona si muove con l'aiuto di una corrente eluente, ciascuno dei componenti separati trascorre una parte del tempo nella fase stazionaria. Quanto più lungo è questo tempo, tanto più lentamente si muove la zona con la miscela da separare. I metodi cromatografici si basano su diversi principi: filtrazione su gel, scambio ionico, adsorbimento, affinità biologica.
Metodo per separare le proteine utilizzando Cromatografia per filtrazione su gel si basa sul fatto che le sostanze di diverso peso molecolare sono distribuite diversamente tra la fase stazionaria e quella mobile. La colonna cromatografica è riempita di granuli di una sostanza porosa ( Sephadex , agarosio, ecc.). Nella struttura del polisaccaride si formano legami incrociati e si formano granuli con “pori”, attraverso i quali passano facilmente acqua e sostanze a basso peso molecolare. A seconda delle condizioni si possono formare granuli con “pori” di dimensioni diverse.
La fase stazionaria è il liquido all'interno dei granuli, nel quale possono penetrare sostanze a basso peso molecolare e proteine a basso peso molecolare. La miscela proteica applicata alla colonna cromatografica viene lavata (eluita) facendo passare un solvente attraverso la colonna. Anche le molecole più grandi si muovono insieme al fronte del solvente.
Le molecole più piccole si diffondono nei granuli di Sephadex ed entrano per qualche tempo nella fase stazionaria, con conseguente ritardo del loro movimento. La dimensione dei pori determina la dimensione delle molecole capaci di penetrare all'interno dei granuli (Fig. 1).
Figura 1. Separazione di una miscela proteica mediante filtrazione su gel.
Poiché la struttura del gel di Sephadex si deforma facilmente sotto pressione, i gel hanno iniziato a essere sostituiti con matrici più rigide (Sefactil, Toy-Operl), che sono granuli sferici con diverse dimensioni dei pori. La scelta delle dimensioni dei pori nei granuli dipende dagli scopi della cromatografia.
Cromatografia a scambio ionico. Proprio come l'elettroforesi, il metodo si basa sulla separazione di proteine che differiscono nella carica totale a determinati valori di pH e forza ionica della soluzione. Quando una soluzione proteica viene fatta passare attraverso una colonna cromatografica riempita con un materiale solido poroso carico, alcune proteine vengono trattenute su di essa a causa delle interazioni elettrostatiche.
Come fase stazionaria vengono utilizzati scambiatori di ioni: sostanze organiche polimeriche contenenti gruppi funzionali carichi.
Esistono scambiatori anionici caricati positivamente, tra i quali il più comunemente usato è la dietilamminoetilcellulosa (DEAE-cellulosa), contenente gruppi cationici, e scambiatori cationici caricati negativamente, ad esempio, la carbossimetilcellulosa (CM-cellulosa), contenente gruppi anionici.
La scelta dello scambiatore ionico è determinata dalla carica della proteina rilasciata. Pertanto, uno scambiatore anionico viene utilizzato per isolare le proteine caricate negativamente. Quando una soluzione proteica viene fatta passare attraverso una colonna, la forza del legame della proteina con lo scambiatore anionico dipende dal numero di gruppi carbossilici caricati negativamente nella molecola. Le proteine adsorbite su uno scambiatore anionico possono essere lavate via (eluite) con soluzioni tampone con diverse concentrazioni saline, molto spesso NaCl, e diversi valori di pH. Gli ioni cloro si legano ai gruppi funzionali caricati positivamente dello scambiatore anionico e spostano i gruppi carbossilici delle proteine. A basse concentrazioni saline, vengono eluite le proteine debolmente legate allo scambiatore anionico. Un aumento graduale della concentrazione di sale o una variazione del pH, che modifica la carica della molecola proteica, porta al rilascio di frazioni proteiche, una delle quali contiene la proteina desiderata.
Cromatografia di affinità o cromatografia di affinità. Questo è il metodo più specifico per isolare singole proteine, basato sull'interazione selettiva delle proteine con ligandi attaccati (immobilizzati) ad un supporto solido. Un substrato o coenzima può essere utilizzato come ligando se si isola un qualsiasi enzima, antigeni per l'isolamento di anticorpi, ecc. Una soluzione contenente una miscela di proteine viene fatta passare attraverso una colonna riempita con un ligando immobilizzato. Al ligando è attaccata solo una proteina che interagisce specificamente con esso; tutte le altre proteine escono con l'eluato (Fig. 2). Le proteine adsorbite sulla colonna possono essere rimosse lavandola con una soluzione con un valore di pH modificato o una forza ionica modificata. In alcuni casi, viene utilizzata una soluzione detergente per rompere i legami idrofobici tra la proteina e il ligando.

Figura 2. Cromatografia di affinità.
La cromatografia di affinità è altamente selettiva e aiuta a purificare la proteina isolata migliaia di volte.
Ultracentrifugazione. Il metodo di separazione si basa anche sulle differenze nei pesi molecolari delle proteine. La velocità di sedimentazione delle sostanze durante la rotazione in un'ultracentrifuga, dove l'accelerazione centrifuga raggiunge 100.000-500.000 g, è proporzionale al loro peso molecolare. Uno strato sottile di una miscela proteica viene applicato sulla superficie della soluzione tampone posta in una cuvetta. La cuvetta viene posizionata nel rotore dell'ultracentrifuga. Quando il rotore ruota per 10-12 ore, le molecole più grandi (con peso molecolare più elevato) si depositano nella soluzione tampone a una velocità maggiore. Di conseguenza, nella cuvetta la miscela proteica viene separata in frazioni separate con diversi pesi molecolari (Fig. 3). Dopo la separazione delle frazioni proteiche, il fondo della cuvetta viene riscaldato con un ago e il contenuto viene raccolto goccia a goccia in piccole porzioni nelle provette.

Figura 3. Cuvetta riempita con una soluzione tampone con frazioni proteiche separate.
Elettroforesi delle proteine. Il metodo si basa sul fatto che ad un certo valore di pH e forza ionica della soluzione, le proteine si muovono in un campo elettrico ad una velocità proporzionale alla loro carica totale. Le proteine con carica netta negativa si muovono verso l'anodo (+), mentre le proteine con carica positiva si muovono verso il catodo (-).
L'elettroforesi viene effettuata su diversi supporti: carta, gel di amido, gel di poliacrilammide, ecc. A differenza dell'elettroforesi su carta, dove la velocità di movimento delle proteine è proporzionale solo alla loro carica totale, in un gel di poliacrilammide la velocità di movimento delle proteine è proporzionale alla loro pesi molecolari.
La risoluzione dell'elettroforesi nel gel di poliacrilammide è maggiore rispetto a quella su carta. Pertanto, durante l'elettroforesi delle proteine del siero umano, sulla carta vengono rilevate solo 5 frazioni principali: albumine, α 1 globuline, α 2 globuline, β-globuline e γ-globuline (Fig. 4). L'elettroforesi delle stesse proteine nel gel di poliacrilammide consente di ottenere fino a 18 frazioni diverse. Per rilevare le frazioni proteiche, strisce di carta o colonne di gel vengono trattate con un colorante (molto spesso blu di bromofenolo o nero ammidico). Il complesso colorato di proteine e colorante rivela la posizione delle varie frazioni sul trasportatore.

Figura 4. Elettroforesi delle proteine del siero del sangue di una persona sana su carta.